Um die Grundlagen der Vibrationsspektroskopie zu verstehen, kann man sich, vereinfacht betrachtet, jedes Molekül als eine Ansammlung aus mehreren Kugeln (den Atomen) und verbindenden, elastischen Federn (den Atombindungen) vorstellen. Entlang jeder Feder können die verbundenen Kugeln schwingen. Die jeweiligen Schwingungen sind von der Feder, den beteiligten Kugeln und dem Winkel zwischen den Federn abhängig und hoch charakteristisch. Die resultierenden Schwingungs-Spektren können im infraroten Spektralbereich in Absorption und mit der Raman-Spektroskopie in Streuung gemessen werden.
Im Gegensatz zu diesem klassischen Model (Kugel-Feder) werden im quantenmechanischen Model diskrete Vibrationsniveaus angeregt. Daher spricht man auch von Vibrationsspektren. In der obigen Abbildung sind die Streckschwingung und Biegeschwingung des Wassermoleküls und die beiden resultierenden IR-Absorptionsbanden gezeigt. Diese beiden Banden sind charakteristisch für ein Wassermolekül. Die resultierenden Spektren sind also charakteristisch für das jeweilige Molekül und vergleichbar mit Fingerabdrücken, anhand derer die Moleküle Label-frei identifiziert werden können.
Bei kleinen Molekülen kann man die einzelnen Banden eindeutig den jeweiligen Schwingungen des Moleküls zuordnen. Bei Proteinen kann man mit Hilfe der FTIR-Differenzspektroskopie die Banden des aktiven Zentrums aus der Hintergrundabsorption des Gesamtproteins selektieren. Die statische FTIR-Differenzspektroskopie wurde von Siebert, Mäntele und Gerwert 1983 (Siebert et al. 1983) eingeführt. Dabei wird ein Intermediat bei tiefen Temperaturen eingefroren. Die einzelnen Differenzbanden konnten zum ersten Mal eindeutig spezifischen molekularen Gruppen, wie Asparaginsäuren mit Hilfe der Isotopenmarkierung (Engelhard, Gerwert et al. 1985 und Gerwert et al. 1986), oder noch präziser, einer einzelnen Asparaginsäure mit Hilfe ortspezifischer Mutagenese (Gerwert et al. 1989), zugeordnet werden.
Dies war der entscheidende Durchbruch der Methode, die anfänglich sehr kritisch gesehen wurde, weil nur sehr kleine Änderungen vor der starken Hintergrundabsorption des gesamten Proteins selektiert werden, ähnlich einer Nadel im Heuhaufen. Ein wichtiger nächster Meilenstein war die Etablierung der zeitaufgelösten FTIR-Differenzspektroskopie, die es erlaubt, zeitaufgelöst simultan die verschiedenen Reaktionen in einem Protein bei Raumtemperatur und damit die Proteindynamik zu messen (Gerwert 1988, Gerwert et al. 1990). Basierend auf der zeitaufgelösten FTIR-Spektroskopie ist es nun möglich, komplementär zur Röntgenstrukturanalyse, die statische Bilder des Grundzustands liefert, Filme der Proteindynamik zu erstellen. Das präzise simultane Starten der Proteinreaktionen ist bei der Differenzspektroskopie entscheidend. Daher ist diese Methode besonders gut für Proteine mit Chromophoren geeignet, weil die Reaktion in diesen Fällen mit einem Laserblitz gestartet werden kann. Durch Einführung von photolabilen, sogenannten "caged"-compounds, wie caged GTP, konnte die Methode auf nicht-chromophore Proteine erweitert werden (Cepus et al. 1998).
Im Gewebe liefert das Vibrationsspektrum einen charakteristischen 'fingerprint', welcher integral das Proteom, Genom, Lipidom und Metabolom reflektiert. Mit Hilfe der Bioinformatik kann man zwischen den unterschiedlichen Mustern unterscheiden.Bei Zellen und Gewebe überlappen so viele Banden der Proteine, der DNA und RNA, der Lipide und weiterer Gruppen im Spektrum, dass man einzelne Banden nicht mehr spezifisch zuordnen kann. Die FTIR-Spektren bilden aber einen "fingerprint", der integral das Proteom und Genom reflektiert. Werden mit Hilfe eines Mikroskops die Spektren ortsaufgelöst aufgenommen und den verschiedenen Spektren dann ortsaufgelöst verschiedene Farben zugeordnet, erhält man ein Label-freies Index-Farben-Bild der Zelle oder des Gewebes. Bei dieser Mustererkennung spielt die bioinformatische Auswertung mit unüberwachten und überwachten Klassifizierern eine zentrale Rolle. Die verschiedenen Farben unterscheiden dann verschiedene molekulare Bereiche. Mit Hilfe der Raman- und CARS-Mikroskopie können somit Zellen Label-frei klassifiziert werden. Mit Hilfe der IR-Mikroskopie kann insbesondere Gewebe Label-frei annotiert werden.
(2) Siebert, F., Mäntele, W., Gerwert, K.
Fourier-transform infrared spectroscopy applied to rhodopsin. The problem of the protonation state of the retinylidene Schiff base reinvestigated
Eur. J. Biochemistry 136, 119-127 (1983).
(4) Engelhard, M., Gerwert, K., Hess, B., Kreutz, W., Siebert. F).
Light-driven protonation changes of internal aspartic acids of bacterio-rhodopsin: An investigation by static and time-resolved infrared difference spectroscopy using [4-13C] aspartic acid labeled purple membrane
Biochemistry 24, 400-407 (1985).
(8) Gerwert, K., Siebert, F.
Evidence for light-induced 13-cis, 14-s-cis, isomerization in bacteriorhodopsin obtained by FTIR difference spectroscopy using isotopically labelled retinals
EMBO Journal 5, 805-811 (1986).
(13) Gerwert, K.
Intramolekulare Proteindynamik untersucht mit zeitaufgelöster Fourier Transform Infrarot-Differenzspektroskopie
Ber. Bunsenges. Phys. Chem. 92, 978-982 (1988).
(19) Gerwert, K., Hess, B., Soppa, J., Oesterhelt, D.
The role of Asp 96 in the proton pump mechanism of bacteriorhodopsin.
Proc. Natl. Acad. Sci. USA 86, 4943-4947 (1989).
Die ATR-FTIR-Spektroskopie ist eine oberflächenbasierte Technik, mit der Protein-Ligand-Interaktionen genau wie bei der weit verbreiteten SPR (surface plasmon resonance) -Spektroskopie Label-frei zeitaufgelöst untersucht werden können. Aufgrund der spektralen Auflösung der Infrarotspektroskopie wird aber zusätzlich die volle molekulare Information der Protein-Ligand-Interaktion zu jedem Zeitpunkt erhalten (Kötting et al. 2012).
Bei der ATR-Technik wird der Infrarotstrahl innerhalb eines Medium mit hohem Brechungsindex, dem internen Reflektionselement (IRE), reflektiert. An der Grenzfläche bildet sich ein sog. evaneszentes Feld aus, das ca. 1 µm in das umgebende Medium hineinreicht und das mit dem auf dem IRE immobilisierten Protein interagiert. Die ATR-Technik ermöglicht damit die Messung von Flüssigkeiten in einem Durchflusssystem im infraroten Spektralbereich.
Durch Ausbildung von Membranmonolagen von natürlichen Lipiden auf dem IRE und die in situ Anbindung von lipidverankertem Ras konnte dieses periphere Membranprotein in einer natürlicheren Umgebung infrarotspektroskopisch untersucht werden. Hierbei zeigte sich, dass membrangebundenes N-Ras eine aufrechte molekulare Orientierung aufweist. Simultane FRET-Untersuchungen zeigten, dass N-Ras membrangebunden als Dimer vorliegt. Dies war Titelgeschichte im Biophysical Journal (Güldenhaupt et al. 2012). Auch Protein-Protein-Interaktion wurde mit dem ATR-System untersucht. So wurde z. B. die Membranextraktion der GTPase Rab durch ein GDI nachgewiesen (Gavriljuk et al. 2013).
Die frei zugängliche ATR-Oberfläche ermöglicht die gezielte Manipulation der immobilisierten Proteine z. B. durch die Zugabe von Liganden. An diesen Monolagen, die nur wenige µg Protein enthalten, können u. a. durch Nutzung von mehrfachen Reflektionen kleinste Konformationsänderungen der immobilisierten Proteine nachgewiesen und molekulare Reaktionsmechanismen untersucht werden (Pinkerneil et al. 2012, Titelgeschichte). Gleichzeitig wird die intrinsische Polarisation des evaneszenten Feldes genutzt, um Orientierungsanalysen von Monolagen von immobilisierten Proteinen und Lipiden durchzuführen.
Die spezifische Immobilisierung von Proteinen auf dem IRE wird in unserem Lehrstuhl über unterschiedliche oberflächenchemische Techniken erreicht, die immer eine schichtweise Modifikation der ATR-Oberfläche unter gleichzeitiger online spektraler Kontrolle zur Grundlage haben. Hierbei wird die erste Schicht durch Molekülmonolagen von Thiolen oder Silanen gebildet, die durch vorherige Aktivierung der Germanium-Oberfläche eine kovalente Bindung zum ATR-Kristall ausbilden (Schartner et al. 2013). Die zweite Schicht ist die Proteininteraktionsschicht, die in der Messzelle mit dem SAM mittels NHS-, Maleimido- oder Click-Chemie gekoppelt wird. Die Proteininteraktionsschicht kann nun aus NTA-Gruppen, Streptavidin oder Antikörpern bestehen, so dass jegliche HisTag-, StrepTag- oder Biotin-tragende und auch jegliche zum verwendeten Antikörper kompatiblen Proteine immobilisiert werden können.
Die so erreichten Oberflächenkonzentrationen der immobilisierten Proteine ermöglichen es, Liganden-induzierte Konformationsänderungen im Zeitbereich von 10 s - 10 h kinetisch aufzulösen (K4DD).
Die Antikörperimmobilisierung erlaubt es zusätzlich, bestimmte krankheitsrelevante Marker-Proteine hochspezifisch aus komplexen Flüssigkeiten (z. B. Blutplasma) zu fangen. Wir haben aufbauend darauf einen Immuno-ATR-Biosensor zur Alzheimerdiagnose entwickelt.
(136) Kötting, C., Güldenhaupt, J., Gerwert, K.
Time-resolved FTIR spectroscopy for monitoring protein dynamics exemplified by functional studies of Ras protein bound to a lipid bilayer
Chemical Physics, 396, 72-83 (2012)
(140) Pinkerneil, P., Güldenhaupt, J., Gerwert, K., Kötting, C.
Surface attached polyhistidine-tag proteins characterized by FTIR difference spectroscopy
ChemPhysChem 2012, 13, 2649 – 2653
(144) Güldenhaupt, J., Rudack, T., Bachler, P., Mann, D., Triola, G., Waldmann, H., Kötting, C. and Gerwert, K.
N-Ras Forms Dimers at POPC Membranes
Biophys. J. 103, 1585-1593 (2012)
Supporting Material
(149) Schartner, J., Güldenhaupt, J., Mei, B., Rögner, M., Muhler, M., Gerwert, K., Kötting, C.
Universal Method for Protein Immobilization on Chemically Functionalized Germanium Investigated by ATR-FTIR Difference Spectroscopy
J. Am. Chem. Soc. 135, 4079-4087 (2013)
(155) Gavriljuk, K., Itzen, A., Goody, R. S., Gerwert, K., Kötting, C.
Membrane extraction of Rab proteins by GDP dissociation inhibitor characterized using attenuated total reflection infrared spectroscopy
Proc. Natl. Acad. Sci., 110, 13380-13385 (2013)
Supporting Material
(174) El-Mashtoly, S.F., Yosef, H.K., Petersen, D., Mavarani, L., Maghnouj, A., Hahn, S.A., Kötting, C., Gerwert, K.
Label-free Raman spectroscopic imaging monitors the integral physiologically relevant drug responses in cancer cells
Anal. Chem. 87 (2015) 7297-7304
Supporting Material
Raman-Mikroskopie ermöglicht eine markierungsfreie chemische Bildgebung. Sie basiert auf der inelastischen Streuung von Photonen an Molekülen. Das Raman-Spektrum eines jeden Moleküls reflektiert die chemische Struktur, da jedes Molekül seine eigene Raman-Signatur (Fingerabdruck) hat.
Spontane Raman-Spektroskopie ermöglicht eine leistungsfähige und zerstörungsfreie Bilderfassung mit hoher molekularer Spezifität und räumlicher Auflösung (einige hundert Nanometer). Die Technik basiert auf der Identifizierung von Molekülschwingungen, die für alle wichtigen Substanzklassen der Zelle charakteristisch sind. Allerdings ist die spontane Raman-Bildgebung relativ langsam, da die Raman-Streuung nur ein sehr kleines Signal liefert. Kohärente Anti-Stokes-Raman-Streuung (CARS) ermöglicht die Verstärkung des schwachen Raman Signals aufgrund nichtlinearer Prozesse. CARS -Bildgebung ist daher schnell und kann mit Videoraten durchgeführt werden.
Wir verwenden diese markierungsfreien Bildgebungstechniken in Kombination mit chemometrischer Analyse zur Bewertung gezielter Krebstherapien und zur Krebsdiagnostik.
Im FTIR-/Raman-Imaging werden ortsaufgelöst Vibrationsspektren aufgenommen. Die Vibrationsspektren repräsentieren dabei die chemische Zusammensetzung des zugrundeliegenden Gewebes. Wir untersuchen humanes Gewebe mit FTIR-/Raman-Imaging. Dabei werden die Spektren als integraler Marker für den biochemischen Status (Proteom, Genom, Transkriptom und Metabolom) des Gewebes zum Zeitpunkt der Entnahme der Probe genutzt. Die Spektren bilden Muster, die charakteristisch für den molekularen Zustand sind, ähnlich wie ein Fingerabdruck charakteristisch für einen Menschen ist.
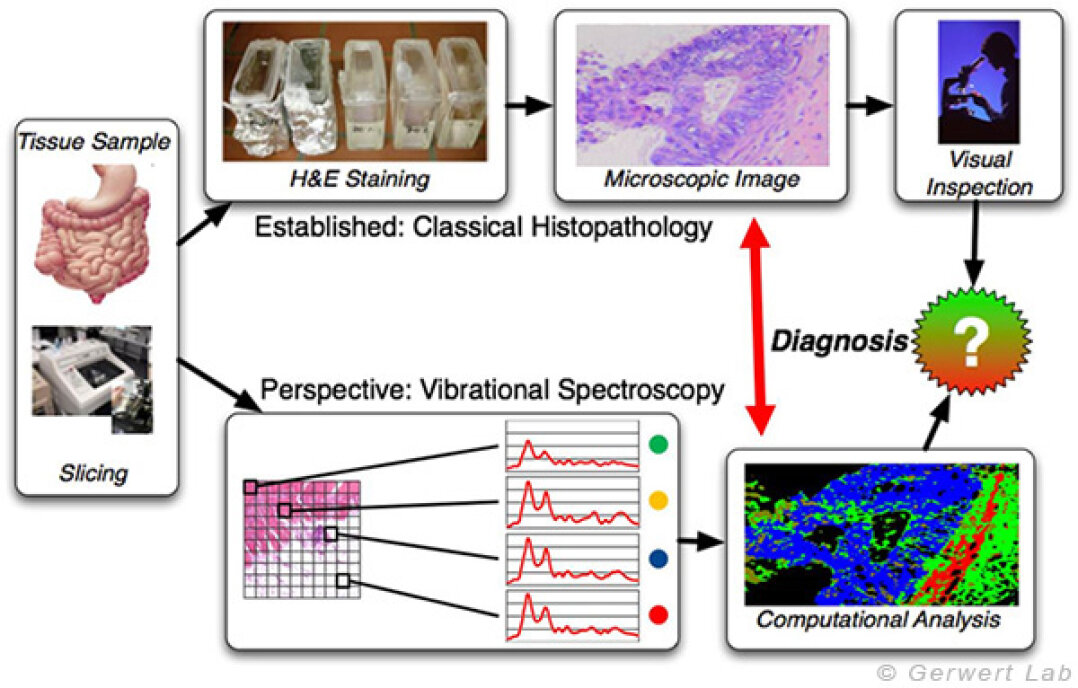
In der Abbildung links ist der Workflow der klassischen Histopathologie und die Klassifikation von Gewebe durch FTIR-/Raman-Imaging gezeigt. Im FTIR/Raman-Imaging wird der Gewebedünnschnitt nicht gefärbt, sondern ortsaufgelöst vermessen. Durch Vergleich mit einer spektralen Datenbank ist eine Label-freie, automatisierte Klassifikation von Gewebe möglich. Der Ansatz ist für Tumorgewebe aus Darm, Lunge und Blase am Lehrstuhl für Biophysik etabliert. Im Vergleich zum Goldstandard in der Klinik, der Histopathologie, erreichen wir eine Sensitivität und Spezifität von über 95% für diese drei Entitäten.
Durchgeführt wird das FTIR-Imaging mit Infrarotmikroskopen, die spezielle FPA-Detektoren (focal plane array) mit 64x64 bzw. 128x128 Elementen nutzen. Dies ermöglicht die simultane ortsaufgelöste Vermessung von großen Gewebeschnitten. Durch weitere technische Modifikationen kann so eine schnelle und kontinuierliche Datengewinnung erfolgen.
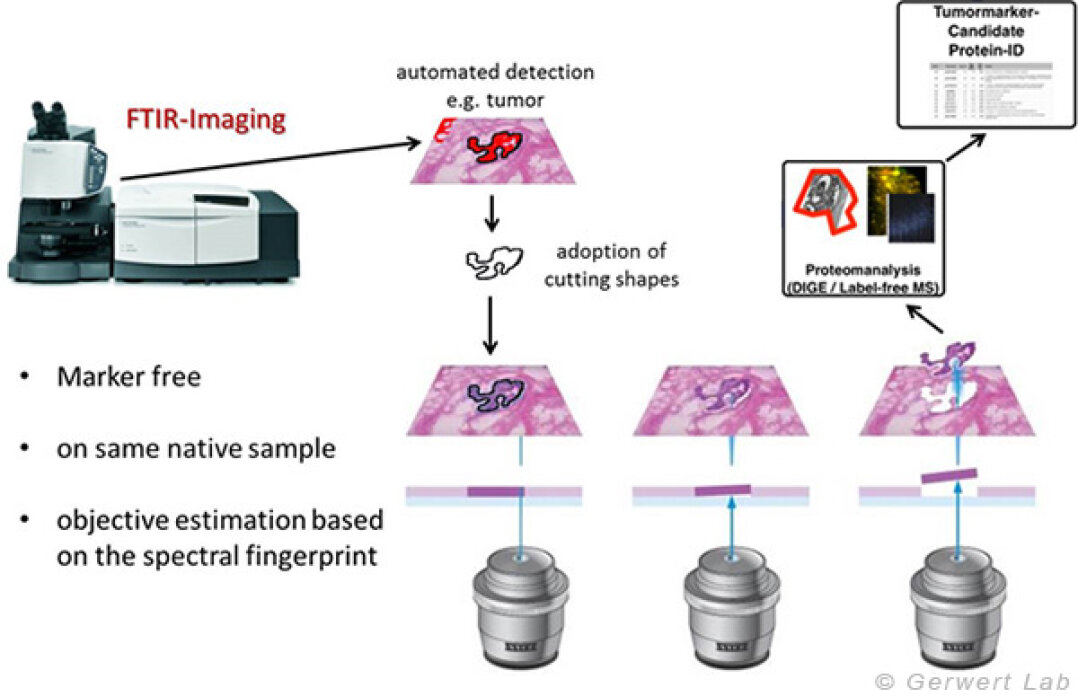
Neben der medizinisch diagnostischen Anwendung kann FTIR-/Raman-Imaging aufgrund der robusten, ortsaufgelösten und beobachterunabhängigen Charakterisierung an unbehandelten Gewebeproben die Biomarkersuche eingesetzt werden. In unserer Gruppe haben wir hierzu ein einzigartiges Verfahren entwickelt, welches die ortsaufgelöste Gewebeerkennung des FTIR-Imagings nutzt, um mit einer Lasermikrodissektion (LMD) Proben für Omics-Techniken zu sammeln. Dies erlaubt es, im Bereich der Biomarkersuche zum ersten Mal ortsaufgelöst an sehr präzise annotierten aber noch unbehandelten Proben zu arbeiten.


Defekte in der Faltung von Proteinen führen zu neurodegenerativen Erkrankungen wie Alzheimer und Parkinson. Daher kann in diesen Fällen der Faltungszustand von Proteinen als Biomarker zur Diagnose von neurodegenerativen Erkrankungen eingesetzt werden. Wir haben einen ATR-basierten Immuno-Sensor für die Alzheimer-Diagnostik entwickelt. Dieser wurde bereits mit Preisen ausgezeichnet. Er ist auf andere neurodegenerative Erkrankungen erweiterbar, insbesondere zur Diagnostik von Parkinson.
Neurodegenerative Erkrankungen
In Deutschland leiden rund 1,5 Millionen Menschen an Demenz. Die häufigsten Demenzformen sind Morbus Alzheimer und die Parkinson Erkrankung. Diesen neurodegenerativen Erkrankungen ist die Fehlfaltung körpereigener Proteine zu β-Faltblatt-reichen Strukturen gemein, die zunächst aggregieren und folglich als sogenannte Plaques (Amyloid-β, Alzheimer), Tangles (Tau) bzw. Lewy Bodies (α-Synuclein, Parkinson) im Gehirn abgelagert werden. Es wird postuliert, dass die Aggregation dieser Markerproteine bereits Jahre vor der klinisch-symptomatischen Manifestation beginnt.
Alzheimer Diagnose an Körperflüssigkeiten (Alzheimer-Sensor)
In der medizinischen Biophotonik ist ein zum Patent angemeldeter immunologischer Infrarot-Sensor entwickelt worden, mit dem erstmals hochspezifisch die Sekundärstrukturverteilung der Amyloid-β- (Aβ) -Fraktion in Liquor- und Blut-Plasma-Proben detektiert werden kann. Im Laufe der Alzheimer-Progression verschiebt sich das Verhältnis von nicht toxischen zu pathologisch auffälligen Aβ-Isoformen, die reich an β-Faltblatt sind, in Körperflüssigkeiten. Die Sekundärstrukturverteilung aller Aβ-Isoformen wird mit dem innovativen ATR-FTIR-Aufbau detektiert und ein spektraler Grenzwert zur Diagnose und Differenzierung von Morbus Alzheimer eingesetzt. Zukünftig wird das immunologisch-sensorische Verfahren auch zur Detektion von Morbus Parkinson angewendet.
Für den Alzheimer-Test in Blut wird eine Genauigkeit von 84%, eine Spezifität von 88% und Sensitivität von 75% erzielt. Erste Ergebnisse zeigen, dass sich der Test zur Früherkennung eignet. Dazu laufen aktuell Forschunsgarbeiten.
Die Projekte stehen im intensiven Kontakt mit Kooperationspartnern aus Forschung und Industrie.
Patent:
K. Gerwert, J. Wiltfang, J. Ollesch, A. Nabers, J. Schartner, and C. Kötting
"Biosensor for Conformation and Secondary Structure Analysis"
WO 2015/121339, 2015.
(172) Nabers, A., Ollesch, J., Schartner, J., Kötting, C., Genius, J., Haußmann, U., Klafki, H., Wiltfang, J., Gerwert, K.
An infrared sensor analysing label-free the secondary structure of the Abeta peptide in presence of complex fluids
J Biophotonics. 2016 Mar;9(3):224-34. doi: 10.1002/jbio.201400145
(178) Nabers, A., Ollesch, J., Schartner, J., Kötting, C., Genius, J., Hafermann, H., Klafki, H., Gerwert, K., Wiltfang, J.
Amyloid-ß-Secondary Structure Distribution in Cerebrospinal Fluid and Blood Measured by an Immuno-Infrared-Sensor: A Biomarker Candidate for Alzheimer's Disease
Anal. Chem. 2016, 88, 2755-2762
Supporting Material
Ziel von Kinetics for Drug Discovery (K4DD) ist es, die Kinetik der Wirkstoff-Protein-Interaktion zu bestimmen, durch die Etablierung, Validierung und Optimierung von einer Reihe von Messprotokollen und Messtechniken die Kinetiken verlässlicher zu messen und die in vivo Wirksamkeit aus der Kinetik vorherzusagen. Das K4DD-Konsortium setzt sich aus 20 Partnern aus 6 europäischen Ländern zusammen. K4DD wird gefördert von der Innovative Medicine Initiative Joint Undertaking (IMI JU). Die komplette Fördersumme beträgt 21 Mio. €.
Es hat sich in den letzten Jahren herausgestellt, dass die Kinetik der Wirkstoff-Protein-Interaktion ein entscheidender Faktor bei der Entwicklung neuer Wirkstoffe ist, insbesondere die koff-Rate, die angibt, wie lange ein Wirkstoff mit dem Zielprotein interagiert. Diese Rate wurde bisher vernachlässigt, scheint aber ein sehr wesentlicher Faktor für die Wirksamkeit zu sein.
Bei der Bindung eines Wirkstoffmoleküls an sein Zielprotein wird bei ca. 30% der Wirkstoffe eine Strukturänderung des Proteins hervorgerufen. Diese Konformationsänderungen führen zu einer geänderten Interaktion des Zielproteins in der Zelle und sind damit maßgeblich für die pharmakologische Wirkung. Da unterschiedliche Wirkstoffe auch unterschiedliche Proteinkonformationen hervorrufen können, ist der Nachweis der Konformationswechselaktivität ein wichtiger Parameter zur Wirkstoffentwicklung, dessen Bestimmung bislang allerdings sehr zeit- und kostenintensiv ist.
Durch Anwendung von ATR-FTIR-Spektroskopie in Kombination mit einer Durchflusszelle sind wir in der Lage, die Konformationswechselaktivität eines Wirkstoffs innerhalb weniger Minuten nachzuweisen und zusätzlich die wichtige koff-Rate zu bestimmen. Möglich ist dies durch die Kombination einer am Lehrstuhl für Biophysik entwickelten Automatisierung des ATR-Aufbaus, mit der die über 10 h dauernden Messabläufe präzise gesteuert werden können und die eigens am Lehrstuhl etablierte oberflächenchemische Veränderung der verwendeten ATR-Kristalle (Schartner et al. 2013, Schartner et al. 2014). Die Kooperationen innerhalb des K4DD-Konsortiums ermöglichen uns den Zugriff auf eine Vielzahl von Zielproteinen, inklusive der dazu gehörigen Inhibitor-Bibliotheken, für die uns auch Röntgenstrukturen der Protein-Inhibitor-Komplexe und kinetische und thermodynamische Daten zu Verfügung gestellt werden. Diese exzellente Basis an Referenzdaten ermöglicht es, die Infrarotdifferenzspektren detailliert zu interpretieren und zusätzlich die extrahierten koff-Raten zu validieren. Die ATR-FTIR-Spektroskopie ist ein wichtiges neues Werkzeug für die Wirkstoffentwicklung.
Der Verband EFPIA vertritt die Interessen von ca. 2200 forschenden Pharmafirmen u.a. den Verband der Forschender Arzneimittelhersteller und viele Einzelunternehmen wie z.B. AstraZeneca, Bayer Healthcare, Boehringer Ingelheim, GlaxoSmithKline, Johnson & Johnson, Merck, Novartis, Pfizer, Recordati Roche, sanofi-aventis u.a.
(149) Schartner, J., Güldenhaupt, J., Mei, B., Rögner, M., Muhler, M., Gerwert, K., Kötting, C.
Universal Method for Protein Immobilization on Chemically Functionalized Germanium Investigated by ATR-FTIR Difference Spectroscopy
J. Am. Chem. Soc. 135, 4079-4087 (2013)
(174) El-Mashtoly, S.F., Yosef, H.K., Petersen, D., Mavarani, L., Maghnouj, A., Hahn, S.A., Kötting, C., Gerwert, K.
Label-free Raman spectroscopic imaging monitors the integral physiologically relevant drug responses in cancer cells
Anal. Chem. 87 (2015) 7297-7304
Supporting Material
Zahlreiche Krankheiten wie Krebs werden durch veränderte Proteininteraktionen in der Zelle verursacht. Das Ziel unserer Projekte auf zellulärer Ebene ist die Entwicklung der Label-freien Raman- und CARS-Mikroskopie in Kombination mit chemometrischen Methoden als innovative Methode für die Zelldiagnostik.
So kann z. B. Blasenkrebs anhand von Zellen aus Körperflüssigkeiten annotiert werden (Stichwort: "Spektrale" Zytopathologie).
Ferner entwickeln wir an Zelllinien in vitro Assays um festzustellen, ob ein bestimmtes Krebsmedikament bei einem Patienten wirksam ist oder nicht. Dieser Ansatz wird zur Label-freien Untersuchung der Pharmakokinetik im Rahmen der "companion diagnostics" genutzt.
Evaluation von gezielten Krebstherapien durch Raman-Mikroskopie
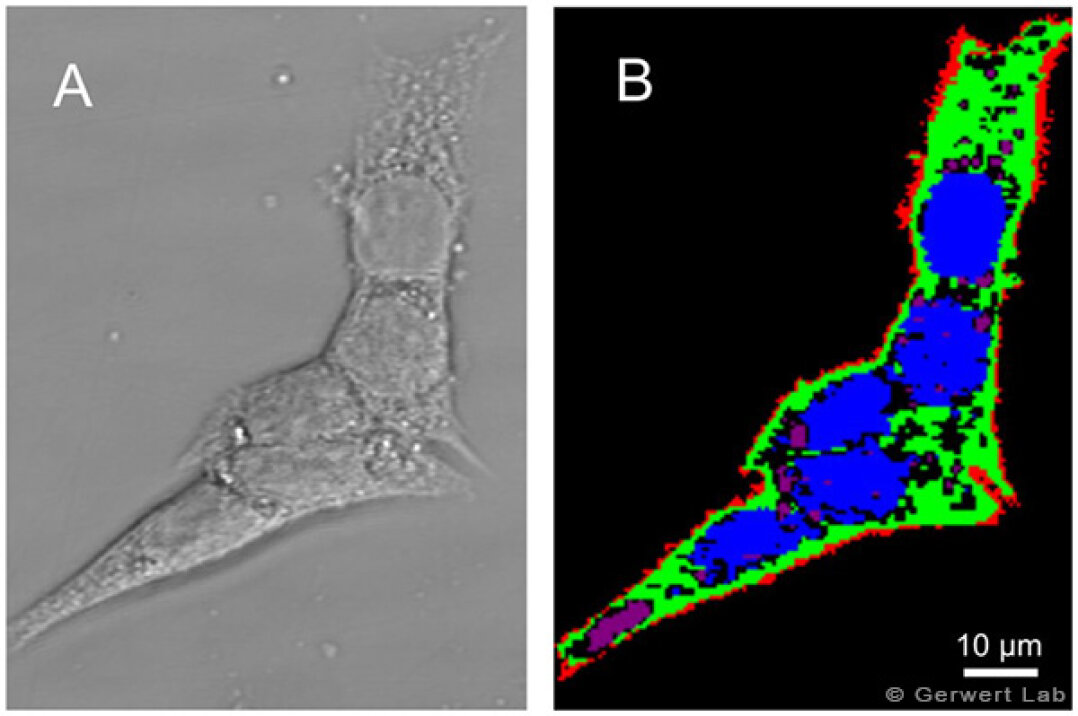
Die Evaluation der Wirksamkeit von Arzneimittelkandidaten in vitro während der frühen Stadien der Medikamentenentwicklung ist unerlässlich. Hierfür können sowohl lineare Raman-Mikroskopie als auch CARS (kohärente anti-Stokes Raman-Streuung)-Mikroskopie als Label-freie und nicht-invasive Screening-Methoden genutzt werden. Zur Etablierung wird eine automatische Erkennung der Zellorganellen durchgeführt. So können Zellkern, Nukleolus, Lipidtropfen, endoplasmatisches Retikulum, Golgi-Apparat und Mitochondrien erkannt werden (Krauß et al. 2015, El-Mashtoly et al. 2014, El-Mashtoly et al. 2015).
Die Raman-Mikroskopie wird zur Detektion der zellulären Antwort auf gezielte Krebstherapien, wie z.B. durch EGFR-Inhibitoren, angewendet. Hierbei kann der Einfluss bestimmter Mutationen der Zelle wie onkogenes K-Ras untersucht werden (El-Mashtoly et al. 2014, Yosef et al. 2015). Außerdem kann die räumliche Verteilung und der Metabolismus von Tyrosinkinase-Inhibitoren in Krebszellen detektiert werden (El-Mashtoly et al. 2014). Somit zeigt die Raman-Mikroskopie ein großes Potential für die Pharmakokinetik in lebenden Zellen.
(160) El-Mashtoly, S.F., Petersen, D., Yosef, H.K., Mosig, A., Reinacher-Schick, A., Kötting, C., Gerwert, K.
Label-free imaging of drug distribution and metabolism in colon cancer cells by Raman microscopy
Analyst 139, 5 (2013) 1155-1161
(161) El-Mashtoly, S.F., Niedieker, D., Petersen, D., Krauss, S.D., Freier, E., Maghnouj, A., Mosig, A., Hahn, S., Kötting, C., Gerwert, K.
Automated Identification of Subcellular Organelles by Coherent Anti-Stokes Raman Scattering
Biophysical Journal 106 (2014), 1910-1920
(171) Krauß, S.D., Petersen, D., Niedieker, D., Fricke, I., Freier, E., El-Mashtoly, S.F., Gerwert, K., Mosig, A.
Colocalization of fluorescence and Raman microscopic images for the identification of subcellular compartments: a validation study
Analyst, 2015 (140) 2360-2368
(174) El-Mashtoly, S.F., Yosef, H.K., Petersen, D., Mavarani, L., Maghnouj, A., Hahn, S.A., Kötting, C., Gerwert, K.
Label-free Raman spectroscopic imaging monitors the integral physiologically relevant drug responses in cancer cells
Anal. Chem. 87 (2015) 7297-7304
Supporting Material
(175) Hesham, K.Y., Mavarani, L., Maghnouj, A., Hahn, S., El-Mashtoly, S.F., Gerwert, K.
In vitro prediction of the efficacy of molecularly targeted cancer therapy by Raman spectral imaging
Anal Bioanal Chem., DOI 10.1007/s00216-015-8875-z
Supporting Material

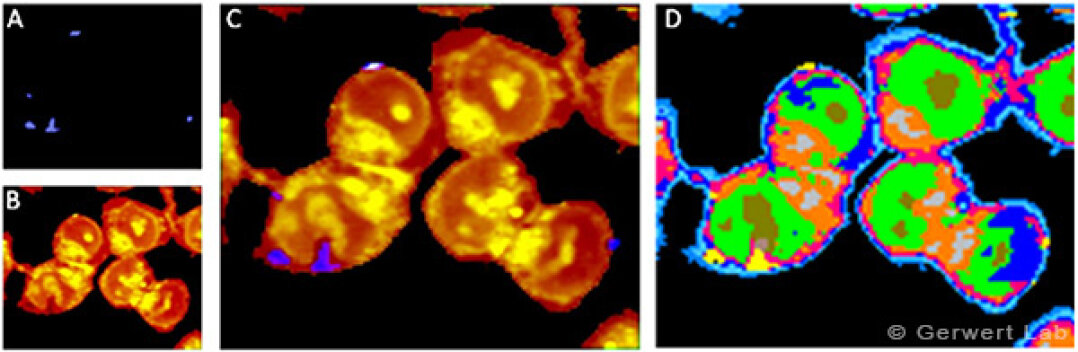

Krebsdiagnose durch Körperflüssigkeiten durch spektrale Raman-Zytopathologie
Die spektrale Zytopathologie (SZP) ist eine nicht-invasive Methode zur Identifikation von Krebszellen in Körperflüssigkeiten wie Urin. Konventionelle zytologische Methoden verwenden die Lichtmikroskopie und sind von der Erkennung morphologischer Strukturen abhängig. Das hat viele Nachteile, so sind ein hoher Zeitaufwand und erfahrene Pathologen nötig, und die meisten Testmethoden haben nur geringe Genauigkeiten. Wir entwickeln eine Raman/CARS-basierte SZP, welche eine schnelle, einfache und reproduzierbare Analyse von Körperflüssigkeiten für die Krebsdiagnose ermöglicht.
Im FTIR-/Raman-Imaging werden ortsaufgelöst Vibrationsspektren aufgenommen. Jedes Spektrum repräsentiert die chemische Zusammensetzung des zugrundeliegenden Gewebes. Wir untersuchen humanes Gewebe mit FTIR-/Raman-Imaging. Dabei werden die Spektren als integraler Marker für den momentanen biochemischen Status (Proteom, Genom, Transkriptom und Metabolom) des Gewebes oder eines Krankheitsstadiums zum Zeitpunkt der Entnahme der Probe genutzt. Die Spektren bilden Muster, die charakteristisch für den molekularen Zustand sind, ähnlich einem Fingerabdruck, der charakteristisch für jeden Menschen ist.
Nachdem eine Gewebeprobe entnommen wurde, wird diese in dünne, mikroskopierbare Scheiben geschnitten (1-10 µm). In der klassischen Histopathologie werden diese nun gefärbt und mit einem Mikroskop betrachtet. Der Pathologe entscheidet dann aufgrund seiner Erfahrung und vorgegebenen Kriterien, welche Diagnose für den Patienten getroffen wird. Im FTIR-Imaging wird der Gewebedünnschnitt nicht gefärbt sondern ortsaufgelöst vermessen. Die erhaltenen Spektren werden bei unbekanntem Gewebe klassifiziert und jeder Klasse eine Farbe zugeordnet. Das erhaltene Indexfarbenbild wird nun mit dem histopathologischen Befund verglichen. Gewebsspezifische Spektren werden anschließend in einer Datenbank gespeichert, die dann zur Charakterisierung unbekannter Gewebeschnitte (überwachte Klassifizierung) genutzt werden kann. Nach erfolgreicher Etablierung einer spektralen Datenbank ist somit eine automatisierte Charakterisierung eines unbekannten Gewebes möglich, welche die Diagnose des Pathologen unterstützen kann.
Durchgeführt wird das FTIR-Imaging mit Infrarotmikroskopen, die spezielle FPA-Detektoren (focal plane array) mit 64x64 bzw. 128x128 Elementen nutzen. Dies ermöglicht die simultane ortsaufgelöste Vermessung von großen Gewebeschnitten. Durch weitere technische Modifikationen kann so eine schnelle und kontinuierliche Datengewinnung erfolgen. In unseren bisherigen Arbeiten haben wir FTIR-Imaging zur hochspezifischen Gewebeerkennung insbesondere an Darm, Blase und Lunge etabliert. In jüngerer Zeit sind die Entitäten Hirn und Muskel hinzugekommen.
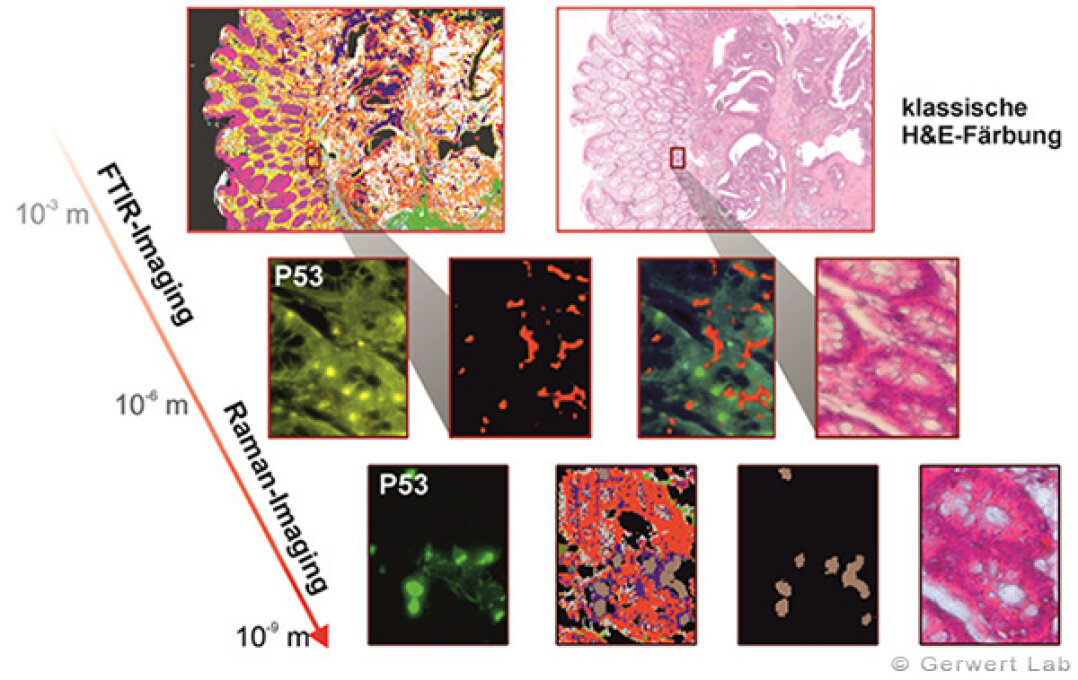
Neben der medizinisch diagnostischen Anwendung kann FTIR-/Raman-Imaging aufgrund der robusten, ortsaufgelösten und beobachterunabhängigen Charakterisierung an unbehandelten Gewebeproben auch zur Biomarkersuche eingesetzt werden. In unserer Gruppe haben wir hierzu ein einzigartiges Verfahren entwickelt, welches die ortsaufgelöste Gewebeerkennung des FTIR-Imagings nutzt, um mit einer Lasermikrodissektion (LMD) Proben für Omics-Techniken zu sammeln. Dies erlaubt es, im Bereich der Biomarkersuche zum ersten Mal an gut charakterisierten unbehandelten Proben zu arbeiten. Erste Ergebnisse sollen in diesem Jahr veröffentlicht werden.




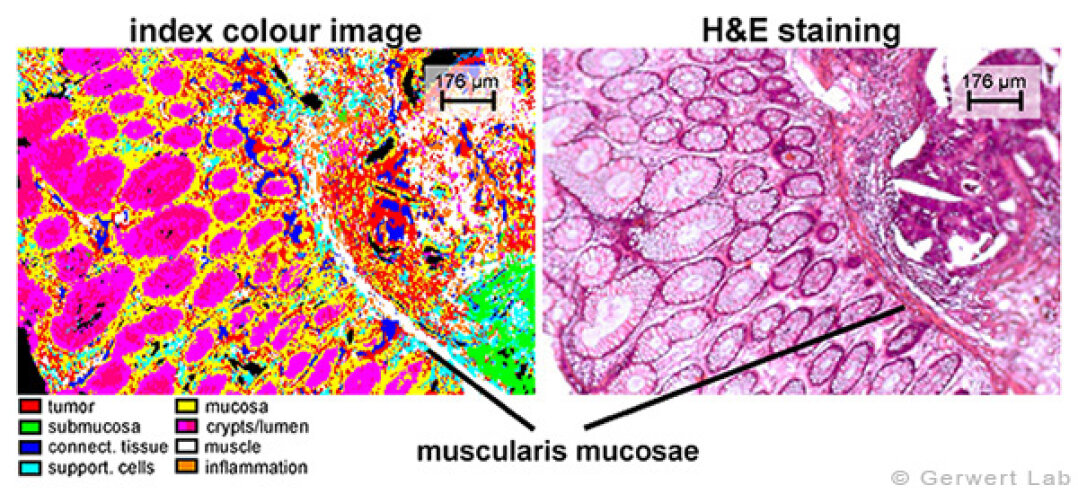
Darmkrebs ist eine der häufigsten Krebserkrankungen in Deutschland, mit steigenden Zahlen aufgrund des immer höheren Altersdurschnitts der Bevölkerung. In Zusammenarbeit mit den Kliniken der Ruhr-Universität Bochum, insbesondere dem Pathologischen Institut in den Projekten PURE und ColoPredict Plus, wird kolorektales Karzinom von uns untersucht. Eine spektrale Datenbank ist angelegt. Mit FTIR- und Raman-Imaging konnte diese Charakterisierung von Gewebedünnschnitten des Darms mit hoher Genauigkeit (>96 %) etabliert werden. Es konnten feinste Details, wie etwa die für eine Einstufung des Karzinoms wichtige muscularis mucosae, aufgelöst werden. Die sehr lokalen Tumorherde sind in der Abbildung in Rot dargestellt. Zum Vergleich ist die später H&E-gefärbte Probe gezeigt.
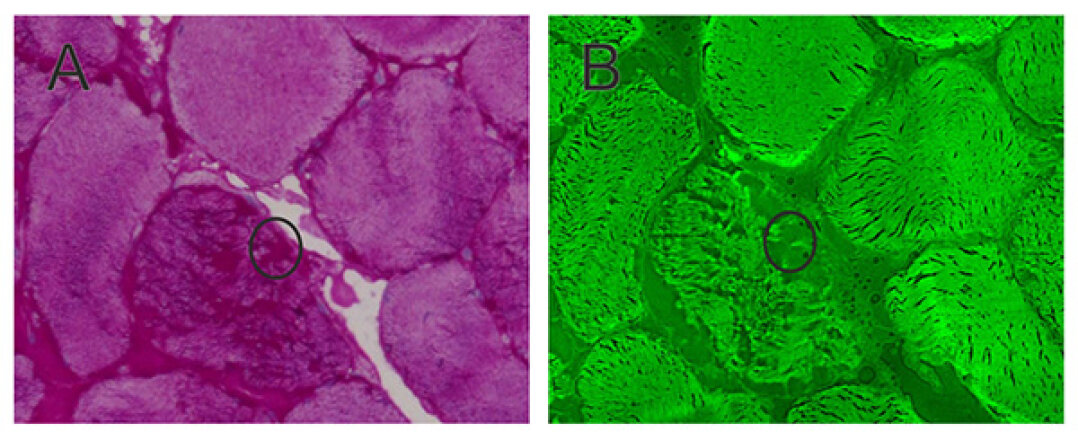
Bei zunehmender Vergrößerung ist die Wellenlängen-begrenzte Auflösung des FTIR-Imagings auf Zellebene deutlich. Durch Kombination mit räumlich höher auflösendem Raman-Imaging können auch Zellstrukturen aufgelöst werden. Dieser Ansatz erlaubt es uns, Fragestellungen, die eine höhere Auflösung erfordern, anzugehen (Abbildung rechts), wie die Erkennung von hoch proliferierenden Zellkernen in eigentlich pathologisch gesunden Krypten (in der Abbildung in Ocker dargestellt).
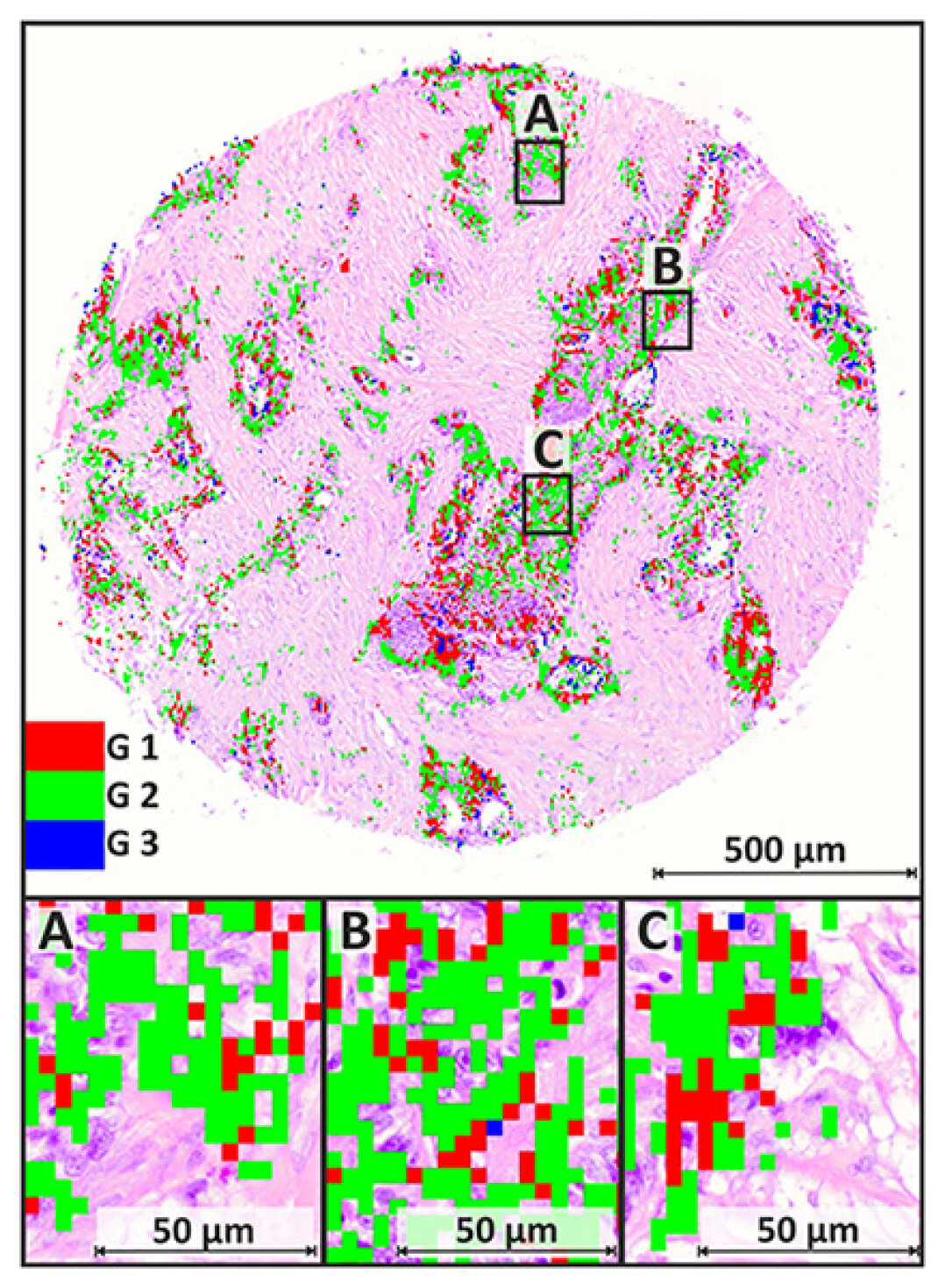
In der modernen Histopathologie stellt sich immer häufiger die Frage nach einer präzisen, differentiellen Diagnostik, die zum einen durch immunohistochemische Verfahren als auch durch biochemische Techniken wie Sequenzierungen unterstützend beantwortet werden können. Mit FTIR-Imaging an Darm war es uns möglich zu zeigen, dass wir die differentielle Fragestellung des Tumorgrades mit Genauigkeiten >89 % beantworten können (siehe Abbildung).
(148) Kallenbach-Thieltges, A., Großerüschkamp, F., Mosig, A., Diem, M., Tannapfel, A., Gerwert, K.
Immunohistochemistry, histopathology and infrared spectral histopathology of colon cancer tissue sections
J. Biophotonics, 6 (1), 88-100 (2013)
(152) Mavarani, L., Petersen, D., El-Mashtoly, S.F., Mosig, A., Tannapfel, A., Kötting, C. and Gerwert, K.
Spectral Histopathology of colon cancer tissue sections by Raman imaging with 532 nm excitation provides label free annotation of lymphocytes, erythrocytes and proliferating nuclei of cancer cells
Analyst, 2013, 138, 4035–4039 (2013)
(180) Kuepper, C., Großerueschkamp, F., Kallenbach-Thieltges, A., Mosig, A., Tannapfel, A., Gerwert, K.
Label-free classification of colon cancer grading using infrared spectral histopathology
Faraday Discussion, DOI 10.1039/C5FD00157A



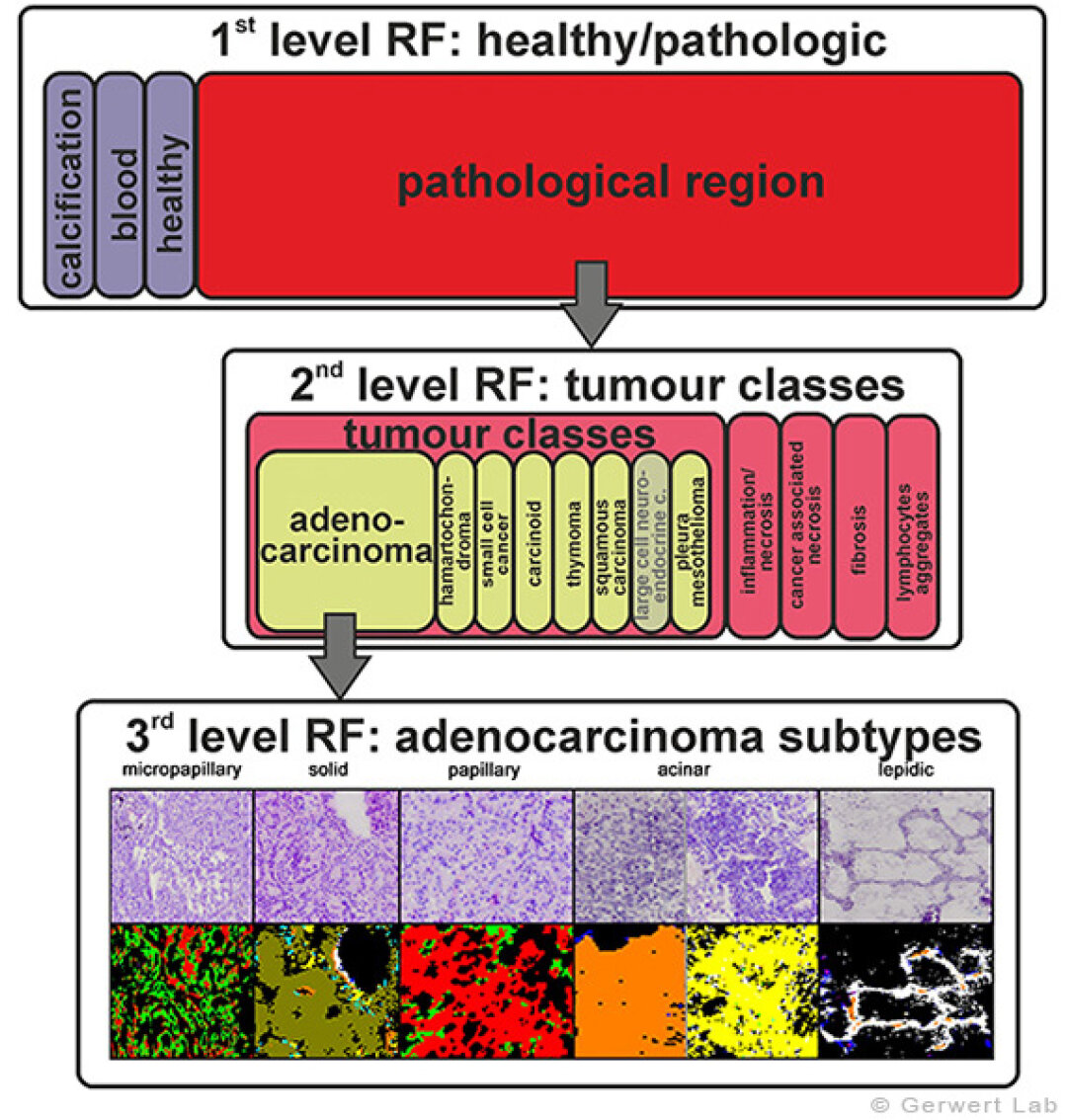
Lungenkrebs ist eine der Krebsarten mit der schlechtesten Prognose für erkrankte Patienten. Dies liegt zum einen an der hohen Komplexität der Erkrankung und zum anderen an der häufig sehr späten Diagnose eines Lungentumors. Lungenkrebs wird in einem Projekt der Deutschen gesetzlichen Unfallversicherung (DGUV) untersucht. Ziel ist es, diagnostische Marker zur Charakterisierung von Lungenkrebs zu finden. Mit FTIR-Imaging war es uns möglich, eine automatisierte Charakterisierung des Lungenkrebses und einwandernder Tumoren (z. B. Mesotheliome der Pleura) mit einer Genauigkeit >94 % zu etablieren. Um den prognostischen Wert dieser Erkennung zu erhöhen, wurde diese um die Erkennung von Tumorsubtypen erweitert. So war es uns als weltweit erster Gruppe möglich, eine automatisierte Erkennung der Subtypen von Adenokarzinomen der Lunge an Gewebedünnschnitten zu etablieren. In absteigender Reihenfolge der Mortalität sind dies mikropapilläres, solides, papilläres, azinäres und ledisches Karzinom. Bisher ist die Zuordnung dieser Subtypen nur über komplizierte immunhistochemische Färbemethoden mit hoher Fehleranfälligkeit möglich.
Aufgrund der hohen Heterogenität von Lungentumoren ist gerade in dieser Entität die Gewinnung von biochemischen Informationen oder gar Biomarkerkandidaten sehr schwierig. Durch unseren Ansatz, der Kombination von FTIR-Imaging mit Lasermikrodissektion zur Probengewinnung für Omics-Techniken, werden wir Proben für die Analyse des Gewebes in bisher nicht erreichter Genauigkeit erhalten. Erste vielversprechende Experimente sind durchgeführt worden und zeigen, dass dieser Ansatz eine sehr präzise molekulare Charakterisierung des Gewebes erlaubt und neue Biomarkerkandidaten liefern wird.
(167) Großerüschkamp, F., Kallenbach-Thieltges, A., Behrens, T., Brüning, T., Altmayer, M., Stamatis, G., Theegarten, D., Gerwert, K.
Marker-free automated histopathological annotation of lung tumour subtypes by FTIR imaging
Analyst, 2015, 140, 2114–2120
(182) Ollesch, J., Theegarten, D., Altmayer M., Darwiche, K., Hager, T., Stamatis, G., Gerwert, K.
An infrared spectroscopic blood test for non-small cell lung carcinoma and subtyping into pulmonary squamous cell carcinoma or adenocarcinoma
Biomedical Spectroscopy and Imaging 5 (2016) 129–144

Blasenkrebs wird in Zusammenarbeit mit den Kliniken der Ruhr-Universität Bochum in den Verbundprojekten PURE und UroFollow untersucht. Im laufenden Projekt konnte mit FTIR-Imaging die automatisierte Charakterisierung von Blasentumoren etabliert werden. Eine zentrale, weiterführende Fragestellung in der Diagnostik von Blasenkrebs ist die differentielle Fragestellung des Tumorgrades. Denn diese entscheidet über die Entnahme der Blase. Dabei ist eine Entzündung der Blase nur sehr schwer von einem high grade Karzinom (carcinoma in situ) zu unterscheiden. Durch Einsatz des FTIR-Imagings ist es uns möglich, eine automatisierte Charakterisierung des Gewebes zu etablieren, die die differentielle Fragestellung des Tumorgrades beantworten kann. Diese sehr präzise und robuste Charakterisierung wird derzeit validiert.
Durch Kombination mit Omics-Techniken soll ein tiefergehendes Verständnis der Biochemie von Blasentumoren gewonnen werden. Vielversprechende Ansätze befinden sich derzeit in der Validierung und versprechen die Entdeckung neuer, präziserer Biomarker.
(159) Ollesch J., Heinze M., Heise H.M., Behrens T., Brüning T., Gerwert K.
It's in your blood: spectral biomarker candidates for urinary bladder cancer from automated FTIR spectroscopy
J. Biophotonics 7, No. 3-4 (2014) 210-221
Author biographies
In diesem neu gestarteten Projekt sollen in Zusammenarbeit mit Herrn Prof. Dr. Matthias Vorgerd (Neurologische Abteilung des Bergmannsheils Bochum) vibrationsspektroskopische Techniken zur Aufklärung der Veränderungen des Muskels durch neurodegenerative Erkrankungen erforscht werden. Hierbei stehen vor allem der Mechanismus der Protein-Aggregation und die markierungsfreie Unterscheidung von Muskeldystrophien im Fokus. Erste Ergebnisse des Raman-, CARS- und FTIR-Imaging zeigen vielversprechende Resultate.