Auf den nachfolgenden Seiten können Sie sich über die aktuell laufenden Projekte am Lehrstuhl für Biophysik informieren.
Unsere Arbeiten fokussieren sich insbesondere auf die beiden Schwerpunkte Molekulare Biophysik, in welchem grundlagenwissenschaftlich orientierte Fragstellungen rund um die molekulare Funktionsweise und Dynamik von Proteinen erforscht werden, und Biospektroskopie, in welchem anwendungsorientierte Fragestellungen, insbesondere neue Ansätze zur (Früh-)Diagnose von Krebs und neurodegenerativen Erkrankungen erforscht werden.
Die medizinisch biophotonischen Arbeiten werden insbesondere im Rahmen der Protein Research Unit Ruhr within Europe (PURE) durchgeführt. Ab 2018 wird der Forschungsbau für molekulare Proteindiagnostik (ProDi) auf dem Gesundheitscampus in Bochum zur Verfügung stehen, in welchem universitäre und klinische Forscher rund um Wissenschaftler des PURE Konsortiums innovative Methoden zur Frühdiagnose von onkologischen und neurodegenerativen Erkrankungen entwickeln werden.
Viele der molekular biophysikalischen Arbeiten werden im Rahmen des SFB 642 durchgeführt.
Wenn Sie mehr erfahren wollen, sprechen Sie uns bitte an.
Im folgenden können Sie sich über den Methodischen Ansatz im thematischen Schwerpunkt Molekulare Biophysik und über unsere aktuellen Arbeiten zum Thema rekombinante Proteine informieren.
Zur Untersuchung der Proteindynamik arbeiten am Lehrstuhl für Biophysik interdisziplinär Physiker, Chemiker, Biochemiker und Biologen eng zusammen.
Im ersten Schritt werden die Proteine mit molekularbiologischen Methoden rekombinant hergestellt und für biophysikalische Untersuchungen aufgereinigt. Im nächsten Schritt wird die dreidimensionale Raumstruktur eines Proteins mit Hilfe der Röntgenstrukturanalyse bestimmt. Die Bestimmung der dreidimensionalen Raumstruktur ist immer ein wichtiger Meilenstein. Es liefert ein Bild des statischen Grundzustands.
Die Proteindynamik wird mit zeitauflösenden spektroskopischen Methoden charakterisiert. Dabei erlauben die am Lehrstuhl für Biophysik weiterentwickelten zeitauflösenden FTIR-spektroskopischen Techniken, molekulare Reaktionsmechanismen von Proteinen mit atomarer Auflösung zu entschlüsseln. Es können die verschiedenen Reaktionen simultan über verschiedene Zeitskalen, von wenigen Nanosekunden bis einigen Sekunden, durch eine Kombination aus Step-Scan-FTIR und Rapid-Scan-FTIR analysiert werden.
Die methodische Weiterentwicklung neuer zeitauflösender infrarotspektroskopischer Methoden zur Analyse der Funktion von Proteinen ist ein wichtiges Ziel am Lehrstuhl für Biophysik. Ergänzt werden die experimentellen Methoden durch biomolekulare Simulationen, Molekulardynamik-Simulationen und quantenchemische QM/MM-Simulationen. Damit können schließlich die in den IR-Spektren kodierten räumlichen Informationen dekodiert werden.
Am Ende hat man dann nicht nur ein statisches Bild des Grundzustands, sondern einen Film von der Dynamik der Proteinfunktion und Proteininteraktion mit atomarer Auflösung.
Basierend auf den Ergebnissen von vielen Veröffentlichungen, konnten schließlich Filme von der lichtgetriebenen Protonenpumpe Bakteriorhodopsin und dem GTPase-Mechanismus des Ras-Proteins erstellt werden. Die Ergebnisse sind in Reviews zusammengefasst.
Am Lehrstuhl für Biophysik werden die untersuchten Proteine rekombinant hergestellt. Dazu werden die hierfür kodierenden Gene kloniert und exprimiert. Im Wesentlichen wird das Escherichia coli-System genutzt. Für die Mikroorganismen-Anzucht stehen eine Reihe von Schüttelinkubatoren, Fermenter im Maßstab von 1 bis 20 Liter Kulturvolumen sowie die erforderliche Infrastruktur für das Downstream-Processing (Kühlzellen, Zentrifugen, Zellaufschlussgeräte) zur Verfügung. Zur biokompatiblen Reinigung von Proteinen sind verschiedene Chromatographie-Apparate (2 Äkta-Purifier und 3 Äkta-Prime) in Betrieb.
Besondere Expertise liegt für die Isolation und Analytik von integralen Membranproteinen vor, insbesondere Licht-, Redox- und ATP-getriebene Ionenpumpen. Neben E. coli werden Proteine und ihre ortsspezifischen Varianten auch im Archaeon Halobacterium salinarum (Bakteriorhodopsin) und in der eukaryotischen Hefe Pichia pastoris (Mikrobielle Retinalproteine) hergestellt.
Außerdem wird für die Kultivierung und Anzucht von Insekten- und mammalischen Zellen im mittleren Maßstab ein separates Zellkulturlabor betrieben.
Wenn Sie mehr über die molekularbiologischen Arbeiten am Lehrstuhl für Biophysik erfahren möchten, lesen Sie bitte hier weiter.
(61) Rammelsberg, R., Huhn, G., Lübben, M., Gerwert, K.
Bacteriorhodopsin's Intramelecular Proton-Release Pathway consists of a Hydrogen-Bonded Network
Biochemistry 37, 5001-5009 (1998)
(63) Lübben, M., Prutsch, A., Mamat, B., Gerwert, K.
Elektron transfer induces side chain conformational changes of Glu-286 from cytochrome bo3
Biochemistry, 38, 2048-2056 (1999)
(127) Brucker, S., Gerwert, K., Kötting, C.
Tyr39 of Ran preserves the Ran·GTP gradient by inhibiting GTP hydrolysis
J. Mol. Biol. 401, 1–6 (2010)
(168e) Kuhne, J., Eisenhauer, K., Ritter, E., Hegemann, P., Gerwert, K., Bartl, F.
Early Formation of the Ion-Conducting Pore in Channelrhodopsin-2
Angew. Chem. Int. Ed. 2015, 54, 4953–4957
Um die 3D-Struktur von Proteinen zu bestimmen, setzen wir vorwiegend die Röntgenkristallographie ein. Hierbei werden zunächst Proteinkristalle gezüchtet (dafür müssen die Proben extrem homogen vorliegen), die dann an unserem Röntgendiffraktometer vermessen werden. Gute Kristalle werden dann zusätzlich an Synchrotronstrahlungsquellen (z. B. in Grenoble, Hamburg oder Villigen) vermessen, um optimale Datenqualität zu erzielen.
Beugungsmuster des Lysozym-KristallsAus diesen Messdaten können wir dann die Elektronendichteverteilung im Kristall bestimmen und damit die Positionen aller Aminosäuren sowie von gebundenen Wassermolekülen oder Liganden. Eine besondere Herausforderung stellen dabei immer noch integrale Membranproteine dar, für die wir spezielle Kristallisationstechniken, wie z. B. Kristallisation in kubischen Lipidphasen, einsetzen.
Im Rahmen unserer Kooperationen mit der AG Röntgenstrukturanalyse an Proteinen (Hofmann) konnten wir so die strukturellen Grundlagen für die Funktion von wichtigen Membranproteinen aufklären. Vor allem die parallele Untersuchung von funktionalen Proteinvarianten mit Röntgenstrukturanalyse und FTIR-Techniken erlaubte uns neue Einblicke in das bakterielle Reaktionszentrum (Hermes et al. 2006) oder Bacteriorhodopsin (Wolf et al. 2010).
Wenn Sie mehr über die Arbeiten der AG Röntgenstrukturanalyse an Proteinen erfahren möchten, lesen Sie bitte hier weiter.
(110) Hermes, S., Stachnik, J.M., Onidas, D., Remy, A., Hofmann, E., Gerwert, K.
Proton uptake in the reaction center mutant L210DN from Rhodobacter sphaeroides via protonated water molecules
Biochemistry (45) 13741-13748 (2006)
Supporting information
(128 deutsch) Wolf, S., Freier, E., Potschies, M., Hofmann, E., Gerwert, K.
Gerichteter Protonentransfer in Membranproteinen mittels protonierter proteingebundener Wassermoleküle: eine Protonendiode
Angew. Chem. 2010, 122, 7041-7046
Der Lehrstuhl für Biophysik ist mit mehreren modernen FTIR-Spektrometern der Firma Bruker (80V, 66Vs) ausgestattet, die durch Eigenentwicklungen des Lehrstuhls für die jeweilige Anwendung optimiert wurden. Die evakuierbaren Strahlengänge der Spektrometer erlauben Rapid-Scan- (Gerwert et al. 1988, Gerwert et al. 1990) und Step-Scan-Messungen (Rammelsberg et al. 1997, Patent: WO1999040413 A1). Ein experimenteller Aufbau ist in der linken Abbildung gezeigt. Damit können in einem Experiment simultan Reaktionen vom Nanosekunden- bis in den Sekundenbereich beobachtet werden.
In der rechten Abbildung sind die IR-Absorptionsänderungen des Channelrhodopsins von 100 ns bis 140 s dargestellt. Die Reaktionen werden durch Lichtblitze von leistungsfähigen Excimer- oder Farbstofflasern gestartet. Chromoproteine können direkt angeregt werden, ansonsten bieten sich photolabile Triggersubstanzen, photocaged compounds wie caged GTP, an. Der Ansatz hochzeitaufgelöster FTIR-Spektroskopie wurde für Bacteriorhodopsin etabliert. Im untenstehenden Film sind zeitgleich die IR-Absorptionsänderungen sowie die zugehörigen Kinetiken entsprechender Markerbanden und im Strukturbild der Protonentransferweg des Proteins dargestellt.
Patent:
Gerwert, K.
"Verfahren zur zeitaufgelösten Messung von Energiespektren molekularer Zustände und Vorrichtung zur Durchführung des Verfahrens"
WO1999040413 A1
(13) Gerwert, K.
Intramolekulare Proteindynamik untersucht mit zeitaufgelöster Fourier Transform Infrarot-Differenzspektroskopie
Ber. Bunsenges. Phys. Chem. 92, 978-982 (1988).
(22) Gerwert, K., Souvignier, G., Hess, B.
Simultaneous monitoring of light-induced changes in protein side-group protonation, chromophore, isomerization, and backbone motion of bacteriorhodopsin by time-resolved Fourier-transform infrared spectroscopy.
Proc. Natl. Acad. Sci. USA, 87, 9774-9778 (1990).
(55) Rink, Th., Riesle, J.,Oesterhelt, D., Gerwert, K., Steinhoff, H.-J.
Spin labeling studies of the conformational changes in the vicinity of D36,D38,T46 and E161 of bacteriorhodopsin during the photocycle
Biophysical Journal 73, 983-993 (1997)
Die ATR-FTIR-Spektroskopie ist eine oberflächenbasierte Technik, mit der Protein-Ligand-Interaktionen genau wie bei der weit verbreiteten SPR (surface plasmon resonance) -Spektroskopie Label-frei zeitaufgelöst untersucht werden können. Aufgrund der spektralen Auflösung der Infrarotspektroskopie wird aber zusätzlich die volle molekulare Information der Protein-Ligand-Interaktion zu jedem Zeitpunkt erhalten (Kötting et al. 2012).
Bei der ATR-Technik wird der Infrarotstrahl innerhalb eines Medium mit hohem Brechungsindex, dem internen Reflektionselement (IRE), reflektiert. An der Grenzfläche bildet sich ein sog. evaneszentes Feld aus, das ca. 1 µm in das umgebende Medium hineinreicht und das mit dem auf dem IRE immobilisierten Protein interagiert. Die ATR-Technik ermöglicht damit die Messung von Flüssigkeiten in einem Durchflusssystem im infraroten Spektralbereich.
Durch Ausbildung von Membranmonolagen von natürlichen Lipiden auf dem IRE und die in situ Anbindung von lipidverankertem Ras konnte dieses periphere Membranprotein in einer natürlicheren Umgebung infrarotspektroskopisch untersucht werden. Hierbei zeigte sich, dass membrangebundenes N-Ras eine aufrechte molekulare Orientierung aufweist. Simultane FRET-Untersuchungen zeigten, dass N-Ras membrangebunden als Dimer vorliegt. Dies war Titelgeschichte im Biophysical Journal (Güldenhaupt et al. 2012). Auch Protein-Protein-Interaktion wurde mit dem ATR-System untersucht. So wurde z. B. die Membranextraktion der GTPase Rab durch ein GDI nachgewiesen (Gavriljuk et al. 2013).
Die frei zugängliche ATR-Oberfläche ermöglicht die gezielte Manipulation der immobilisierten Proteine z. B. durch die Zugabe von Liganden. An diesen Monolagen, die nur wenige µg Protein enthalten, können u. a. durch Nutzung von mehrfachen Reflektionen kleinste Konformationsänderungen der immobilisierten Proteine nachgewiesen und molekulare Reaktionsmechanismen untersucht werden (Pinkerneil et al. 2012, Titelgeschichte). Gleichzeitig wird die intrinsische Polarisation des evaneszenten Feldes genutzt, um Orientierungsanalysen von Monolagen von immobilisierten Proteinen und Lipiden durchzuführen.
Die spezifische Immobilisierung von Proteinen auf dem IRE wird in unserem Lehrstuhl über unterschiedliche oberflächenchemische Techniken erreicht, die immer eine schichtweise Modifikation der ATR-Oberfläche unter gleichzeitiger online spektraler Kontrolle zur Grundlage haben. Hierbei wird die erste Schicht durch Molekülmonolagen von Thiolen oder Silanen gebildet, die durch vorherige Aktivierung der Germanium-Oberfläche eine kovalente Bindung zum ATR-Kristall ausbilden (Schartner et al. 2013). Die zweite Schicht ist die Proteininteraktionsschicht, die in der Messzelle mit dem SAM mittels NHS-, Maleimido- oder Click-Chemie gekoppelt wird. Die Proteininteraktionsschicht kann nun aus NTA-Gruppen, Streptavidin oder Antikörpern bestehen, so dass jegliche HisTag-, StrepTag- oder Biotin-tragende und auch jegliche zum verwendeten Antikörper kompatiblen Proteine immobilisiert werden können.
Die so erreichten Oberflächenkonzentrationen der immobilisierten Proteine ermöglichen es, Liganden-induzierte Konformationsänderungen im Zeitbereich von 10 s - 10 h kinetisch aufzulösen (K4DD).
Die Antikörperimmobilisierung erlaubt es zusätzlich, bestimmte krankheitsrelevante Marker-Proteine hochspezifisch aus komplexen Flüssigkeiten (z. B. Blutplasma) zu fangen. Wir haben aufbauend darauf einen Immuno-ATR-Biosensor zur Alzheimerdiagnose entwickelt.
(136) Kötting, C., Güldenhaupt, J., Gerwert, K.
Time-resolved FTIR spectroscopy for monitoring protein dynamics exemplified by functional studies of Ras protein bound to a lipid bilayer
Chemical Physics, 396, 72-83 (2012)
(140) Pinkerneil, P., Güldenhaupt, J., Gerwert, K., Kötting, C.
Surface attached polyhistidine-tag proteins characterized by FTIR difference spectroscopy
ChemPhysChem 2012, 13, 2649 – 2653
(144) Güldenhaupt, J., Rudack, T., Bachler, P., Mann, D., Triola, G., Waldmann, H., Kötting, C. and Gerwert, K.
N-Ras Forms Dimers at POPC Membranes
Biophys. J. 103, 1585-1593 (2012)
Supporting Material
(149) Schartner, J., Güldenhaupt, J., Mei, B., Rögner, M., Muhler, M., Gerwert, K., Kötting, C.
Universal Method for Protein Immobilization on Chemically Functionalized Germanium Investigated by ATR-FTIR Difference Spectroscopy
J. Am. Chem. Soc. 135, 4079-4087 (2013)
(155) Gavriljuk, K., Itzen, A., Goody, R. S., Gerwert, K., Kötting, C.
Membrane extraction of Rab proteins by GDP dissociation inhibitor characterized using attenuated total reflection infrared spectroscopy
Proc. Natl. Acad. Sci., 110, 13380-13385 (2013)
Supporting Material
(174) El-Mashtoly, S.F., Yosef, H.K., Petersen, D., Mavarani, L., Maghnouj, A., Hahn, S.A., Kötting, C., Gerwert, K.
Label-free Raman spectroscopic imaging monitors the integral physiologically relevant drug responses in cancer cells
Anal. Chem. 87 (2015) 7297-7304
Supporting Material
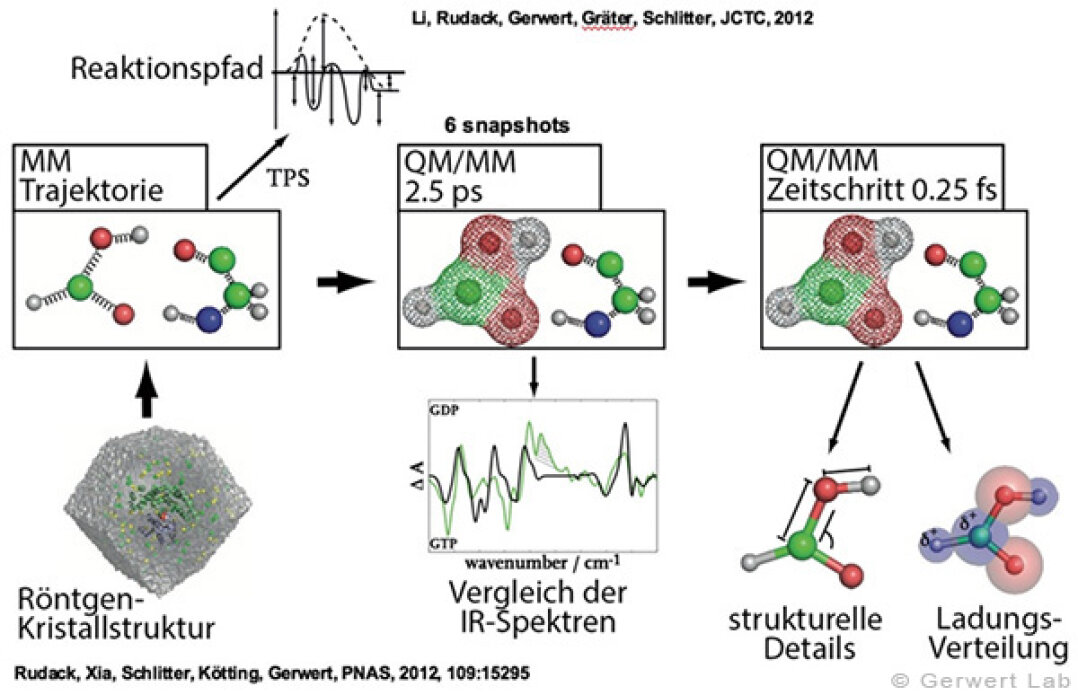
Am Lehrstuhl für Biophysik werden neben experimentellen Methoden ebenfalls computergestützte biomolekulare Simulationen von Makromolekülen und Proteinen durchgeführt. Dazu zählen vor allem molekulardynamik- (MD-) Simulationen, in denen Atome mittels klassischer Mechanik betrachtet werden, und quantenmechanische (QM-) Simulationen, in denen ebenfalls die Verteilung von Elektronen berechnet wird. Wir verfügen dazu über einen eigenen leistungsfähigen Computercluster (hpcpure - 1400 CPUs) und diverse GPU-gestützte Systeme, mit denen das Verhalten von Proteinen und Makromolekülen mit subatomarer Genauigkeit bei hoher Zeitauflösung (fs) über lange Zeiträume (µs) berechnet werden kann.
Durch die Berechnung von IR-Spektren (theoretische IR-Spektroskopie) ist eine enge Verzahnung mit den Experimenten am Lehrstuhl möglich. So können die Berechnungen einerseits verifiziert werden, andererseits experimentelle Befunde erklärt und so neue molekulare Mechanismen entschlüsselt werden. Dazu gehört z. B. die Berechnung von FTIR-Spektren in GTPasen (Rudack et al. 2012), die Untersuchungen des Hydrolysemechanismus in Ras ermöglicht (Rudack et al. 2012), Untersuchungen wie einzelne Wasserstoffbrücken in heterotrimeren G-Proteinen den Nukleotidaustausch beeinflussen (Schröter et al. 2015), Simulationen, die Mechanismen von Transmembranproteinen wie Channelrhodopsin aufklären (Eisenhauer et al. 2012) bis hin zur makromolekularen Organisation von Ras-Dimeren auf Membranen (Güldenhaupt et al. 2012).
(138) Eisenhauer K., Kuhne J., Ritter E., Berndt, A., Wolf S., Freier E., Bartl F., Hegemann P., Gerwert K.
In channelrhodopsin-2 E90 is crucial for ion selectivity and is deprotonated during the photocycle
Journal of Biological Chemistry, 287 (9), 6904-6911 (2012)
(142) Rudack, T., Fei X., Schlitter, J., Kötting, C., Gerwert, K.
Ras and GTPase-activating protein (GAP) drive GTP into a precatalytic state as revealed by combining FTIR and biomolecular simulations
Proc. Natl. Acad. Sci., 109, 15295-15300 (2012)
(143) Rudack, T., Fei X., Schlitter, J., Kötting, C., Gerwert, K.
The role of magnesium for geometry and charge in GTP hydrolysis, revealed by QM/MM simulations
Biophysical Journal, July 2012 (103), 293-302
Supporting Material
(144) Güldenhaupt, J., Rudack, T., Bachler, P., Mann, D., Triola, G., Waldmann, H., Kötting, C. and Gerwert, K.
N-Ras Forms Dimers at POPC Membranes
Biophys. J. 103, 1585-1593 (2012)
Supporting Material
(172) Schröter, G., Mann, D., Kötting, C., Gerwert, K.
Integration of Fourier Transform Infrared Spectroscopy, Fluorescence Spectroscopy, Steady-State Kinetics and Molecular Dynamics Simulations of Gαi1 distinguishes between the GTP Hydrolysis and GDP Release Mechanism
J. Biol. Chem. (2015) 290 (28) 17085-17095

Mit Hilfe der unter Methodischer Ansatz beschriebenen Methoden untersuchen wir die Reaktionsmechanismen und Interaktionen von rekombinanten Membranproteinen (Bakteriorhodopsin, Channelrhodopsin, photosynthetisches Reaktionszentrum) sowie von kleinen und heterotrimeren GTPasen, die mit Membranankern an Lipid-Membranen gebunden sind.
Bakteriorhodopsin (bR) ist eine lichtgetriebene Protonenpumpe. Es ist ein wichtiges biophysikalisches Modellsystem, welches weltweit von vielen Arbeitsgruppen intensiv erforscht wurde. Im Zuge der Untersuchungen wurden viele moderne biophysikalische Methoden neu entwickelt; z. B. die zeitaufgelöste FTIR-Differenzspektroskopie (Gerwert 1988). Heute wird das bR in der Optogenetik zum Abschalten neuronaler Prozesse eingesetzt.
Protonentransfer durch das bR-Protein: Rolle Protein-gebundener Wasser auf der Abgabe und der Aufnahme SeiteDokumentiert in vielen Veröffentlichungen, haben wir den Protonentransferweg im Detail auflösen können (Gerwert et al. 2013). Nach Licht-induzierter all-trans- zu 13-cis-Isomerisierung des Chromophors Retinal, wird die zentrale Protonenbindestelle, die Schiff'sche Base, deprotoniert. Über die protonierte Schiff'sche Base ist das Retinal kovalent am Lys216 des bR gebunden. Durch die Isomerisierung wird der pK-Wert der Schiff'schen Base erniedrigt und Asp85 im L nach M Übergang protoniert (1). Dies konnten wir zum ersten Mal mit Hilfe der zeitaufgelösten FTIR-Differenzspektroskopie nachweisen (Gerwert et al. 1989; Gerwert et al. 1990).
Die Protonierung von Asp85 induziert eine Abwärtsbewegung des Arg82 (2). Dies haben wir in pK-Wert-Berechnungen vorhergesagt (Bashford, Gerwert 1992) und experimentell nachgewiesen (Wolf et al. 2010). Auf der Abgabe-Seite konnten wir zum ersten Mal in einem Protein einen protonierten Wassercluster identifizieren (Garczarek, Gerwert 2006). Dieser stellt die Protonenabgabegruppe dar. Anders als in den von dem Nobelpreisträger Manfred Eigen identifizierten, protonierten Wasserclustern in reinem Wasser spielen die umgebenden Aminosäuren, insbesondere Glu198, Glu204 und Arg82, eine zentrale Rolle bei der Ladungsstabilisierung (Wolf et al. 2014).
Das Zurücksetzen der Pumpe erfolgt über Asp96. Im M nach N Übergang (4) wird Asp96 deprotoniert und reprotoniert die Schiff'sche Base. Die Reprotonierung der Schiff'schen Base durch Deprotonierung von Asp96 konnten wir zum ersten Mal nachweisen (Gerwert et al. 1990). Damit haben wird die zentralen Schritte der Pumpe, die Protonenabgabe an Asp85 und die Protonenaufnahme von Asp96, zum ersten Mal aufgelöst (Gerwert et al. 1989; Gerwert et al. 1990). Basierend auf diesen Ergebnissen ist ein erstes räumlich aufgelöstes Protonenpumpmodell vorgeschlagen worden, das heute Eingang in die Lehrbücher gefunden hat (Gerwert 1992).
Für diesen Protonentransferweg von Asp96 zur Schiff'schen Base werden drei Wassermoleküle durch eine gezielte Konformationsänderung des Proteins zu einer transienten Grotthuß-Kette arrangiert. Diese Grotthuß-Kette leitet dann das Proton von Asp96 zur Schiff'schen Base. Wir konnten zum ersten Mal eine solche transiente Grotthuß-Kette in einem Protein nachweisen (Freier et al. 2011). Asp96 wir von der zytoplasmatischen Seite reprotoniert. (6) Nach Rückisomeriserung zu all-trans-Retinal wird Asp85 im letzten Schritt von O nach BR deprotoniert und somit die Abgabegruppe reprotoniert (Gerwert et al. 1990).
Damit konnten wir zum ersten Mal im Detail die Rolle funktionaler Wassermoleküle in einem Protein auflösen. Diese Ergebnisse sind in einem Übersichtsartikel zusammengefasst (Gerwert et al. 2013).
Der Reaktionszyklus von bR
nach: Garczarek, F., Gerwert, K. Nature (439) 109-112 (2006)
bR - The Movie
nach: Gerwert, K., Freier, E., Wolf, S., Biochim. Biophys. Acta 1837 (2014) 606–613
QM-MM Simulation von proteingebundenen Wassern
nach: Gerwert, K., Freier, E., Wolf, S., Biochim. Biophys. Acta 1837 (2014) 606–613
protonierte Wassercluster: Aufsicht
nach: Garczarek, F., Gerwert, K.Nature (439) 109-112 (2006)
transiente (1ms) protonenleitende Wasserkette
nach: Freier, E., Wolf, S., Gerwert, K. PNAS 108 (28), 11435-11439 (2011)
Simulation der transienten Wasserkette mit QM/MM
nach: Wolf, S., Freier, E., Cui, Q., Gerwert, K., J Chem Phys 2014, 141(22), 22D524
Channelrhodopsin ist ein lichtaktivierbarer Ionenkanal, mit dem sich in der Optogenetik gezielt und mit hoher räumlicher und zeitlicher Auflösung insbesondere neuronale Prozesse untersuchen lassen.
Channelrhodopsin gehört, wie Bakteriorhodopsin, zu den mikrobiellen Rhodopsinen. Mit dem gleichen methodischen Ansatz, den wir für Bakteriorhodopsin etabliert haben, konnten wir zusammen mit Peter Hegemann das frühe Öffnen des Kanals zeitlich und räumlich auflösen (Eisenhauer et al. 2012, Kuhne et al. 2015). Insbesondere war die Kombination aus Step-Scan-FTIR-Spektroskopie, Homologie-Modelling und das Simulieren der Dynamik proteingebundener Wassermoleküle der Schlüssel zum Erfolg. Den wesentlichen Unterschied zur Protonenpumpe Bakteriorhodopsin macht die Helix 2, mit einer Vielzahl von Glutamaten, insbesondere Glu90.
Wir konnten zeigen, dass die Retinal-Isomerisierung zu einer Destabilisierung von Glu90 führt und diese Gruppe nach unten klappt und deprotoniert (linke Abbildung). Dadurch wird die Pore geöffnet (rechte Abbildung), Wasser dringt in das hydrophobe Vestibül oberhalb der Pore ein und drückt Helix 2 nach außen, so dass der Kanal sich öffnet (Abb. unten).
Dieses sogenannte EHT-Model kann das Verhalten verschiedener ortspezifischer Mutanten erklären und bildet jetzt die Basis für das Design optimal angepasster optogenetischer tools. Diese Arbeit war 2015 Titelgeschichte in "Angewandte Chemie".
(138) Eisenhauer K., Kuhne J., Ritter E., Berndt, A., Wolf S., Freier E., Bartl F., Hegemann P., Gerwert K.
In channelrhodopsin-2 E90 is crucial for ion selectivity and is deprotonated during the photocycle
Journal of Biological Chemistry, 287 (9), 6904-6911 (2012)
(168g) Kuhne, J., Eisenhauer, K., Ritter, E., Hegemann, P., Gerwert, K., Bartl, F.
Die frühe Entstehung der ionenleitenden Pore in Channelrhodopsin-2
Angew. Chem. 2015, 127, 5037-5041
Kleine GTPasen sind molekulare Schalter, die zahlreiche Prozesse in Zellen regulieren. Am intensivsten wird das Ras-Protein untersucht, welches das Signal für das Zellwachstum in Richtung Zellkern schaltet. Onkogene Punktmutationen von Ras in der GTP-Bindestelle verhindern die GTP-Hydrolyse von GTP zu GDP. Durch diese Hydrolysereaktion wird die GTPase normalerweise ausgeschaltet und die Wachstumssignalkette unterbrochen. In der obenstehenden Abbildung ist das am gamma-Phosphat angreifende Wasser gezeigt. Diese Reaktion ist in Lösung sehr langsam (200 Tage bei 25°C) wird in Ras aber um 5 Größenordnungen beschleunigt (20 min). Durch GTPase-aktivierende Proteine (GAPs) wird eine Beschleunigung um 5 weitere Größenordnungen erreicht (100 ms) und damit das Zellwachstum kontrolliert. Bei onkogenen Mutationen wird diese Katalyse inhibiert und unkontrolliert ein permanentes Wachstumssignal erzeugt. Dies trägt zur Krebsentstehung bei.
Den Katalyse-Mechanismus konnten wir durch die Kombination von experimenteller und theoretischer Infrarotspektroskopie im Detail aufklären: So konnten wir zum ersten Mal die Bewegung der wichtigsten Aminosäure des GAPs, dem Argininfinger, in die katalytische Bindetasche zeitaufgelöst verfolgen und zeigen, dass dieser Prozess entropiegetrieben ist (Kötting et al. 2008). Die Bewegung des Argininfingers induziert eine Ladungsverschiebung in Richtung Produktzustand (Rudack et al. 2012). Weiterhin wird GTP durch Proteinbindung, ähnlich einer Stahlfeder bei einem Spielzeugauto, in Richtung Übergangszustand gespannt. Damit wird der Bindungsbruch erleichtert. Die Ergebnisse sind in dem untenstehenden Film zusammengefasst.
Mit diesem Ansatz haben wir weitere kleine GTPasen untersucht. Eukaryotische Zellen besitzen 100-150 verschiedene kleine GTPasen. Allen GTPasen gemeinsam ist die G-Domäne. Unterschiede ergeben sich bei der Dynamik des Aufbaus des katalytischen Zentrums, bestehend aus G-Domäne und dem komplementärem GAP. So bildet sich bei den Ras-Gap- (Kötting et al. 2006) und Rap-GAP-Komplexen (Chakrabarti et al. 2007) während der GTP-Hydrolyse ein Intermediat, bei dem der Bindungsbruch bereits vollzogen, das Phosphat aber noch stabil im Proteinkomplex gebunden ist. Somit ist die Abgabe des Phosphats aus dem Protein der geschwindigkeitsbestimmende Schritt. Unsere Charakterisierung des Intermediates war 2006 Titelgeschichte in PNAS (Kötting et al. 2006).
Bei den GTPasen RhoA, Ran (Brucker et al. 2010) und Rab1 (Gavriljuk et al. 2012) mit ihren jeweiligen GAPs ist hingegen der Bindungsbruch geschwindigkeitsbestimmend. Unterschiede ergeben sich ebenfalls beim sogenannten Argininfinger, der für die meisten GTPasen katalytisch entscheidend ist (Ras, Rho, Rab), in anderen aber gar nicht vorkommt (Ran). Gemeinsam ist allen GTPasen ein Säureamid, welches in einigen Fällen eine intrinsische Aminosäure der GTPase ist, in anderen aber durch ein GAP bereitgestellt wird. Dies konnten wir insbesondere für das Rab herausarbeiten (Gavriljuk et al. 2012). Der dynamische Aufbau des katalytischen Zentrums bestimmt die hohe Spezifität zwischen der jeweiligen kleinen GTPase und dem zugehörigen GAP.
Für die biologische Funktion der meisten GTPasen ist die Bindung an die Membran wichtig, an die sie über Lipidanker gebunden sind. Man unterscheidet z. B. zwischen H-, N- und K-Ras. Das K-Ras spielt eine zentrale Rolle bei der Krebsentstehung. Wir konnten zum ersten Mal zeigen, dass Ras an Membranen dimerisiert (Güldenhaupt et al. 2012). Dieser Mechanismus ermöglicht jetzt einen völlig neuen Angriffspunkt für Medikamente. Unsere bisherigen Arbeiten an kleinen GTPasen haben wir in einem Übersichtsartikel zusammengefasst (Kötting, Gerwert 2015). Die Dimerisierung des Ras, der Einfluss auf die Wachstumssignalkette und die Intervention der Dimerisierung mit kleinen potentiellen Wirkstoffen wird derzeit bei uns im Labor intensiv untersucht. Ebenfalls wird der nukleophile Angriff des Wassermoleküls und die Entwicklung potentieller Wirkstoffe erforscht.
(108) Kötting, C., Blessenohl, M., Suveyzdis, Y., Goody, R.S., Wittinghofer, A., Gerwert, K.
A phosphoryl transfer intermediate in the GTPase reaction of Ras in complex with its GTPase-activating protein
Proc. Natl. Acad. Sci USA (103) 13911-13916 (2006)
(111) Chakrabarti, P., Daumke, O., Suveyzdis, Y., Kötting, C., Gerwert, K., Wittinghofer, A.
Insight into catalysis of a unique GTPase reaction by a combined biochemical and FTIR approach
J. Mol. Biol. (367) 383-995 (2007)
(117) Kötting, C., Kallenbach, A., Suveyzdis, Y., Wittinghofer, A. Gerwert, K.
The GAP arginine finger movement into the catalytic site of Ras increases the activation entropy
Proc. Natl. Acad. Sci, 105, 6260-6265 (2008)
(127) Brucker, S., Gerwert, K., Kötting, C.
Tyr39 of Ran preserves the Ran·GTP gradient by inhibiting GTP hydrolysis
J. Mol. Biol. 401, 1–6 (2010)
(143) Rudack, T., Fei X., Schlitter, J., Kötting, C., Gerwert, K.
The role of magnesium for geometry and charge in GTP hydrolysis, revealed by QM/MM simulations
Biophysical Journal, July 2012 (103), 293-302
Supporting Material
(144) Güldenhaupt, J., Rudack, T., Bachler, P., Mann, D., Triola, G., Waldmann, H., Kötting, C. and Gerwert, K.
N-Ras Forms Dimers at POPC Membranes
Biophys. J. 103, 1585-1593 (2012)
Supporting Material
(147)Gavriljuk, K., Gazdag, E.-M., Itzen, A., Kötting, C., Goody, R.S., Gerwert, K.
Catalytic mechanism of a mammalian Rab·RabGAP complex in atomic detail
Proc. Natl. Acad. Sci., 109, 21348-21353 (2012)
(169) Kötting, C., Gerwert, K.
Review: What vibrations tell us about GTPases
Biol. Chem. 2015; 396(2): 131–144
Das Interaktionsnetzwerk von Ras
nach: Henrik te Heesen
Heterotrimere GTPasen haben einen den kleinen GTPasen ähnlichen Schaltmechanismus. Wir untersuchen insbesondere das Ausschalten der Gα-Untereinheiten, das für die Regulation zahlreicher Signaltransduktionswege entscheidend ist. Fehlfunktionen von Gα-Proteinen sind Grundlage vieler Krankheiten wie z. B. fibrösen Displasien, Krebs, Keuchhusten und Cholera. Die wichtigste Methode ist hierbei die zeitaufgelöste FTIR-Spektroskopie, mit der wir die an der Reaktion beteiligten Aminosäuren, wie z. B. den intrinsischen Argininfinger und das katalytische Glutamin, verfolgen können, um ihre genaue Funktion zu bestimmen. Mit der gleichen Technik untersuchen wir auch den Mechanismus von Toxinen, die Gα-Proteine modifizieren. Die Kombination der experimentellen Ergebnisse mit biomolekularen Simulationen ermöglicht die Identifikation weiterer Details des Reaktionsmechanismus mit höchster Auflösung.
(101) Kötting, C., Gerwert, K.
Proteins in action monitored by Time-resolved FTIR Spectroscopy
Chem Phys. Chem (6) 1-8 (2005)
(169) Kötting, C., Gerwert, K.
Review: What vibrations tell us about GTPases
Biol. Chem. 2015; 396(2): 131–144
(172) Schröter, G., Mann, D., Kötting, C., Gerwert, K.
Integration of Fourier Transform Infrared Spectroscopy, Fluorescence Spectroscopy, Steady-State Kinetics and Molecular Dynamics Simulations of Gαi1 distinguishes between the GTP Hydrolysis and GDP Release Mechanism
J. Biol. Chem. (2015) 290 (28) 17085-17095